摘要
当我们十多年前开始研究人工智能 (AI) 时, 很多人怀疑, 在可预见的未来, 这项技术能否能发展到产出有用成果的程度。 但我们始终相信, AI 有潜力造福人类。我们通过训练 AI 系统玩象棋、围棋和 Atari 等游戏, 使其变得更聪明、更有能力。2016 年, 我们利用这些智能系统, 尝试解决一个已经困扰科学界 50 年的生物学基本问题——蛋白质折叠问题。这就是 AlphaFold 诞生的过程。它是一种AI 系统, 可以根据蛋白质的氨基酸序列预测其三维结构。在本文中, 你将了解 AlphaFold 的成就, 这些成就展示了AI 加速科学发现并造福社会的强大能力。
德米斯 ⋅ 哈萨比斯 (Dr. Demis Hassabis) 和约翰 ⋅ 琼珀 (Dr. John Jumper) 因开发了 AlphaFold 系统而获得 2023 年加拿大盖尔德纳国际奖。这一 AI 解决方案被认为解决了困扰科学界 50 年的蛋白质结构预测难题。AlphaFold 被用于构建人类蛋白质组最完整且最精确的图谱——人类体内所有蛋白质的集合, 这具有巨大潜力推动生物学和医学研究的进展。
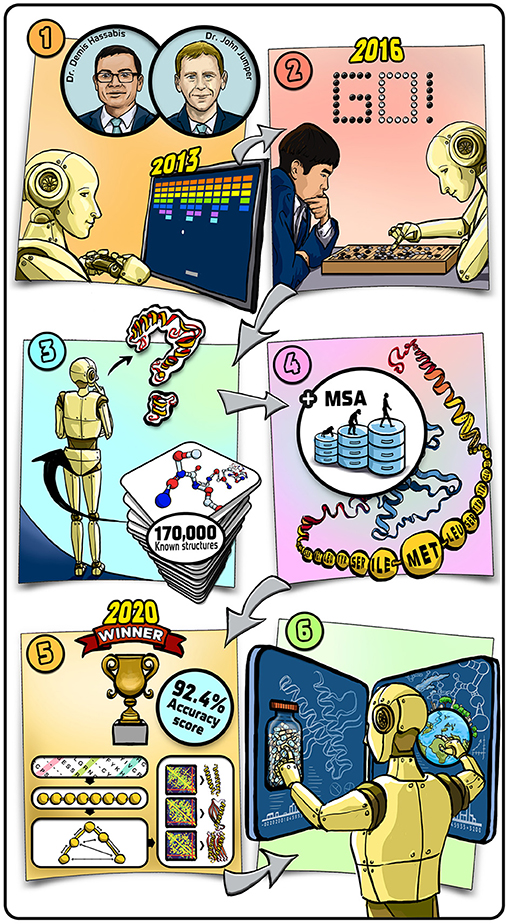
- 图文摘要 - (1) 2013 年开始, 我们训练 AI 系统通关经典的电脑游戏。
- (2) 接着, 我们让 AI 与真人进行更复杂的游戏对战, 并在 2016 年, 我们的系统在围棋比赛中战胜了当时的世界冠军。
- (3) 不久之后, 我们开始着手解决蛋白质折叠问题, 并训练AI系统学习已知的蛋白质结构。
- (4) 为了进一步训练系统, 我们引入了包含蛋白质进化信息的数据库。
- (5) 到 2020 年, 我们的系统在预测蛋白质三维结构时的平均准确率达到了 92.4%。
- (6) 我们希望这一系统能帮助开发新药物, 解决气候变化问题, 并帮助科学家理解这些参与生命构造的微小机器。
小小的生命机器
你知道吗?我们体内几乎所有的过程都是由一种叫做蛋白质的小型生物机器完成的。蛋白质帮助我们视物、移动、消化食物、对抗疾病, 以及完成许多其他维持我们生存和健康的基本动作 (观看视频了解更多关于蛋白质的信息)。目前, 科学界已经发现了超过 2 亿种蛋白质, 并且新蛋白质不断被发现。
蛋白质是由叫做氨基酸的“小积木”构成的 (观看视频了解更多关于蛋白质及其组成的信息)。你可以把蛋白质想象成一串珠子, 其中氨基酸就是那些珠子。蛋白质是在细胞内的“工厂”—— 核糖体中制造的 (你可以从这篇诺贝尔奖合辑文章中了解更多关于核糖体的知识)。在核糖体中, DNA 中的遗传密码会被翻译成氨基酸链。然后, 令人惊叹的事情发生了——这些氨基酸链会折叠成复杂的三维结构, 这些结构决定了蛋白质能够执行的功能。
50 年来的问题
自 20 世纪 60 年代初以来, 科学家们一直在试图理解氨基酸链的特定序列是如何形成蛋白质的特定三维结构的。这就是所谓的蛋白质折叠问题 [1]。由于蛋白质对生物体至关重要, 蛋白质折叠问题被认为是生物化学中最重要的问题之一。研究任何蛋白质时, 科学家可以很容易地确定该蛋白质含有哪些氨基酸, 甚至可以精确排列氨基酸的顺序。但是, 多年来一直难以确定氨基酸链折叠成的最终三维形状, 而这些形状才是形成有效蛋白质的关键。毕竟, 蛋白质的体积太小, 无法用显微镜直接观察其形状。
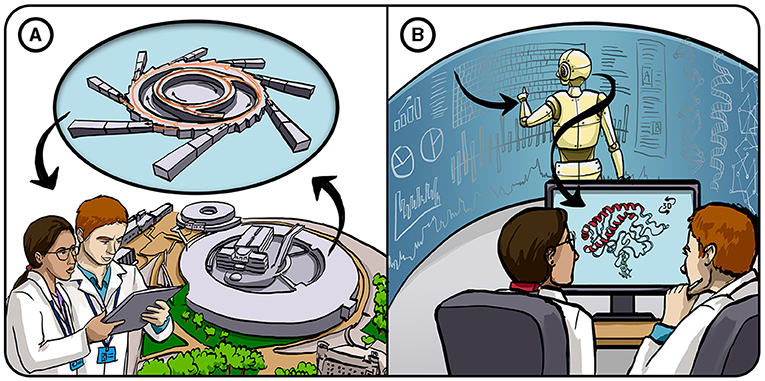
为了弄清蛋白质的三维结构, 科学家们传统上使用了一种叫做 X 射线晶体学的技术 (图 1)。这涉及将蛋白质结晶, 即将许多相同的蛋白质“ 冻结”成重复的三维图案。然后使用大型机器将高能 X 射线射向结晶的蛋白质(图 1A), 最后, 研究人员通过分析 X 射线产生的图案并进行复杂的数学运算来解释结果, 从而确定蛋白质的实际结构。每种蛋白质的这一过程可能需要数年时间!过去 50 年间, 使用诸如 X 射线晶体学、冷冻电子显微镜 (点击这里阅读更多关于冷冻电子显微镜的知识) 和核磁共振分析等方法, 已经确定了大约 20 万种蛋白质的结构, 这些结构数据被存放在公开数据库: 蛋白质银行 中。
- 图 1 - 解决蛋白质折叠问题。
- (A) 传统上, 蛋白质的结构是通过实验确定的, 这些实验利用大型昂贵的设备将X 射线打在晶体化的蛋白质上 (X射线晶体学), 然后通过复杂的数学方法解释结果。 (B) 我们在 DeepMind 的做法是利用先进的人工智能系统, 使用已知的蛋白质结构和蛋白质数据库, 学习预测尚未经过实验测试的蛋白质结构。此方法节省了大量时间和资源。
尽管这个过程很成功, 但显然速度太慢且成本过高, 尤其是当我们想要找出超过 2 亿种已知蛋白质的结构时, 这个数字是我们目前已确定结构的 1000 多倍!
那么, 为什么确定蛋白质的最终三维形状如此困难呢?就像一根鞋带一样, 氨基酸链可能有无数种折叠方式。即使是由 150 个氨基酸组成的小蛋白质, 其可能的折叠构型也多达 10∧300 种 (10∧300 是 1 后面有 300 个零——比宇宙中的恒星还多!)。有如此多的可能折叠方式, 科学家如何在不进行诸如 X 射线晶体学那样耗时且昂贵的实验的情况下知道哪一种是正确的呢?
因此, 在谷歌 DeepMind, 我们决定利用人工智能的力量解决蛋白质折叠问题:计算机从示例中学习并发展出足以解决复杂问题的能力。这种方法已被证明非常有效, 节省了大量时间、金钱和人力, 同时还为我们提供了有关蛋白质工作方式的新见解(图 1B)。
从游戏获胜到解决科学问题
我们在谷歌 DeepMind 的方法是将我们对人工智能的热情与对科学的热情相结合, 寻找人工智能帮助人类的方法。起初, 我们通过教计算机游戏规则并让其通过经验进行改进, 教会了我们的系统如何玩简单的计算机游戏。我们的下一个目标是让这些系统赢得更复杂的游戏, 作为解决现实世界难题的跳板。这包括训练一个 AI 模型去玩围棋, 这是一种非常复杂的棋类游戏, 可能的棋盘配置超过 10∧170 种(比已知宇宙中的原子还多!)。几年间, 我们在游戏情境中开发并测试了 AI 系统, 以观察其表现并不断训练其变得更强大。2016 年, 我们的系统之一 AlphaGo 击败了围棋世界冠军李世石——这一成就此前被认为是不可想象的。这是一个重要的里程碑, 证明我们的 AI 系统足够智能, 能够应对复杂问题。
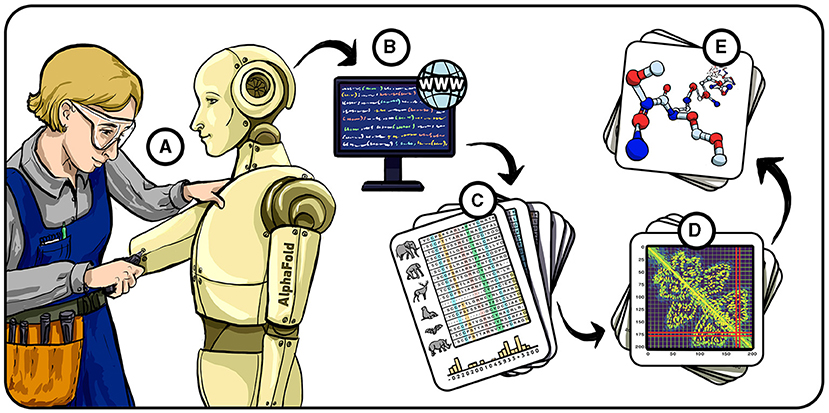
谷歌 DeepMind 在科学研究领域有着自豪的根基, 因此蛋白质折叠问题对我们来说是自然而然的下一步 (图 2)。在 AlphaGo 于 2016 年取得成就后不久, 我们组建了一个团队, 开始致力于根据蛋白质的氨基酸序列预测其结构。这个新的 AI 系统被称为 AlphaFold (图 2A)。AlphaFold 的设计目的是从公开数据库 (如蛋白质数据银行) 中发布的蛋白质结构信息中学习。总体上, 我们可以使用大约 17 万个已知蛋白质结构来训练我们的 AI 系统。我们设计的 AlphaFold 采用类似于人脑处理信息的方式, 使用计算机科学中的一种概念, 叫做人工神经网络 (想了解更多关于人工神经网络和机器学习的内容, 可以阅读Frontiers for Young Minds 的这篇文章)。像人脑一样, AlphaFold 可以通过经验学习并提升其表现。我们提供给它的蛋白质结构例子越多, 它在预测新蛋白质结构时的表现就越好。
- 图 2 - 预测蛋白质折叠的阶段。
- (A) 2016 年, 我们开始构建 AlphaFold——一个解决蛋白质折叠问题的 AI 系统。 (B) AlphaFold 使用来自蛋白质数据库的信息训练自己, 从氨基酸序列中预测蛋白质的三维结构。 (C) 我们还使用了 MSA (多个序列比对) 训练 AlphaFold, MSA 是基于不同生物体中具有相似功能的蛋白质的氨基酸序列集合, 这些蛋白质应该具有相似的结构。那些在序列之间“共同进化”的氨基酸 (彩色列) 携带着关于哪些氨基酸在三维结构中可能接近的重要信息。 (D) 使用这些输入信息, AlphaFold 预测每两个氨基酸之间的距离和角度。 (E) 最后, AlphaFold 将这些距离和角度转换为蛋白质的三维结构预测。
然而, 即便有 17 万个例子, 仍然不足以达到我们所追求的高水平性能——我们需要更多信息来训练 AlphaFold。因此, 我们使用了包含蛋白质序列的开放数据库 (图 2B) 来构建我们所称的多序列比对 (MSA, 图 2C)。MSA 包含与 AlphaFold 要预测的蛋白质在进化上相关的序列, 这些序列一同包含关于结构的线索。蛋白质的形状决定了它们能执行的功能, 许多生物体必须执行相同的生物功能, 如在血液中携带氧气。这意味着不同生物体中所有携氧蛋白质的三维结构在进化过程中可能保持相似, 即使它们的基础氨基酸序列发生了变化。为了实现这一点, 意味着每当一个氨基酸在蛋白质中某个位置发生变化, 另一个与它在三维结构中最接近的氨基酸也必须相应地变化, 以维持原有的形状。我们称这种现象为氨基酸的共同进化, 通过将这些信息输入到 AlphaFold 中, 我们让系统能够检测到氨基酸之间的隐藏关系。
一旦我们向 AlphaFold 输入了足够的信息, 系统就能够预测关于蛋白质形状的基本信息, 包括每两个氨基酸之间的距离 (图 2D) 和角度, 以及预测的可信度 (即预测的可靠性)。这些信息在系统内多次“循环”使用, 每一轮 AlphaFold 都会改进其预测。最后, 它使用关于蛋白质形状的基本概念来预测蛋白质结构中每个原子的三维位置 (图 2E)。在一开始, 我们测试了 AlphaFold 对已知结构的蛋白质的预测, 并让 AlphaFold 通过从错误中学习不断改进自己, 直到其预测变得更准确。经过训练后, 我们使用相同的网络来对未知结构进行预测。
AlphaFold 的进化
在 AlphaFold 的旅程中, 一个令人激动的里程碑发生在 2018 年, 当时 AlphaFold 在两年一度的蛋白质结构预测竞赛 CASP 中获得了第一名。AlphaFold 在最难的蛋白质上得到了约 60 分 (满分 100 分) [2], 这比之前的最佳得分(约 40 分)有了巨大的飞跃。这让我们对 AlphaFold 的能力更加充满信心, 并决定在下次评估中进一步改进系统。在我们下一版的系统 AlphaFold 2 中, 我们将更多关于氨基酸链物理和几何的科学知识整合进了系统的学习过程中, 并将其与我们对蛋白质折叠问题的理解结合起来。基本上, 我们教会了 AlphaFold 2 如何进行 MSA 分析, 然后利用改进的 MSA 分析更好地理解蛋白质折叠 (从而理解氨基酸链的物理和几何特性)。这种信息的往返流动提升了 AlphaFold 2 的性能。
在 2020 年的 CASP14 结构预测竞赛中, AlphaFold 2 以惊人的 92.4 分 (满分 100 分) 赢得了比赛 [3]。这几乎接近通过实验 (如X射线晶体学) 确定蛋白质结构的准确性, 但没有高时间成本和费用。因此, AlphaFold 2 被公认为解决了一个 50 年未解的蛋白质折叠问题 (见CASP14 新闻稿)。
尽管这是一个伟大的成就, 但这仅仅是开始。2020 年, 我们发布了约 33 万个蛋白质的预测结构, 到 2022 年, 这一数字已超过 2 亿。随着时间的推移, 我们从这些结构中获得的知识将使我们更好地理解蛋白质生物学以及蛋白质在细胞中的协作工作。这一能力将帮助许多人, 从开发新药和疫苗, 到通过设计新的食塑酶应对气候变化 [4, 5]。 像 AlphaFold 2 这样的 AI 系统还可以加速科学发现的进程。想象一下, 如果我们利用 AI 系统的强大学习能力来解决各个科学和工程领域中的难题, 科学的发展速度将会有多快。这是一个非常激动人心的时代, 我们鼓励大家保持关注, 与我们一同踏上利用 AI 解开世界最有趣谜团的旅程!
术语表
蛋白质 (Proteins): ↑ 微小的生物机器, 负责在我们体内执行大部分功能。
氨基酸 (Amino acids): ↑ 构成蛋白质的基本单元。
蛋白质折叠问题 (Protein-folding problem): ↑ 1960 年代提出的一个科学问题, 探讨如何根据蛋白质的氨基酸序列预测其三维结构。
X射线晶体学 (X-ray crystallography): ↑ 一种利用X射线确定蛋白质三维结构的实验方法。
人工智能 (Artificial intelligence): ↑ 计算机像人脑一样学习并模仿人类智能的能力。
多序列比对(MSA) (Multiple sequence alignments (MSAs)): ↑ 来自不同生物体的蛋白质的氨基酸序列, 这些蛋白质由于具有相似的功能, 应该具有相似的结构。
利益冲突声明
DH 和 JJ 受雇于 Google DeepMind。
扩展阅读
- 2023 加拿大盖尔德纳国际奖获奖者: Demis Hassabis 博士、 John Jumper 博士.
- Demis Hassabis 博士、 John Jumper 博士——2023 加拿大盖尔德纳国际奖 (YouTube).
- 蛋白质折叠问题解决了吗?——Sabine Hossenfelder (YouTube).
- DeepMind——首页.
致谢
感谢诺亚 ⋅ 塞格夫 (Noa Segev) 为本文基础进行采访并共同撰写文章, 感谢伊里斯 ⋅ 加特 (Iris Gat )提供图表。
参考文献
[1] ↑ Dill, K. A., Ozkan, S. B., Shell, M. S., and Weikl, T. R. 2008. The protein folding problem. Annu. Rev. Biophys. 37:289–316. doi: 10.1146/annurev.biophys.37.092707.153558
[2] ↑ Senior, A. W., Evans, R., Jumper, J., Kirkpatrick, J., Sifre, L., Green, T., et al. 2019. Protein structure prediction using multiple deep neural networks in the 13th Critical Assessment of Protein Structure Prediction (CASP13). Proteins 87:1141–8. doi: 10.1002/prot.25834
[3] ↑ Jumper, J., Evans, R., Pritzel, A., Green, T., Figurnov, M., Ronneberger, O., et al. 2021. Applying and improving AlphaFold at CASP14. Proteins 89:1711–21. doi: 10.1002/prot.26257
[4] ↑ Thornton, J. M., Laskowski, R. A., and Borkakoti, N. 2021. AlphaFold heralds a data-driven revolution in biology and medicine. Nat. Med. 27:1666–9. doi: 10.1038/s41591-021-01533-0
[5] ↑ Callaway, E. 2022. What’s next for the AI protein-folding revolution. Nature 604:234–8. doi: 10.1038/d41586-022-00997-5
