摘要
几十年前, 科学家们都认为细菌是非常简单的生物, 彼此不会交流, 只擅长增殖。近来, 我们发现真相绝非如此!细菌通过一种名为“群体感应”的语言进行交流, 这就像世界上最早的社交网络!本文将介绍群体感应的发现过程, 以及它如何彻底改变了我们对微生物世界的理解。我们还将探讨这项新知识如何帮助医生治疗致命细菌感染。让我们一起探索有趣的细菌语言, 看看这种语言如何帮助人类保持健康。
Bonnie Bassler、Michael Silverman 和 E. Peter Greenberg 三位博士发现了细菌之间及其与周围非细菌细胞的通信机制, 由此荣获 2023 年加拿大盖尔德纳国际奖。他们的研究揭示了微生物的行为模式, 为开发抗感染新药开辟了激动人心的新方向。
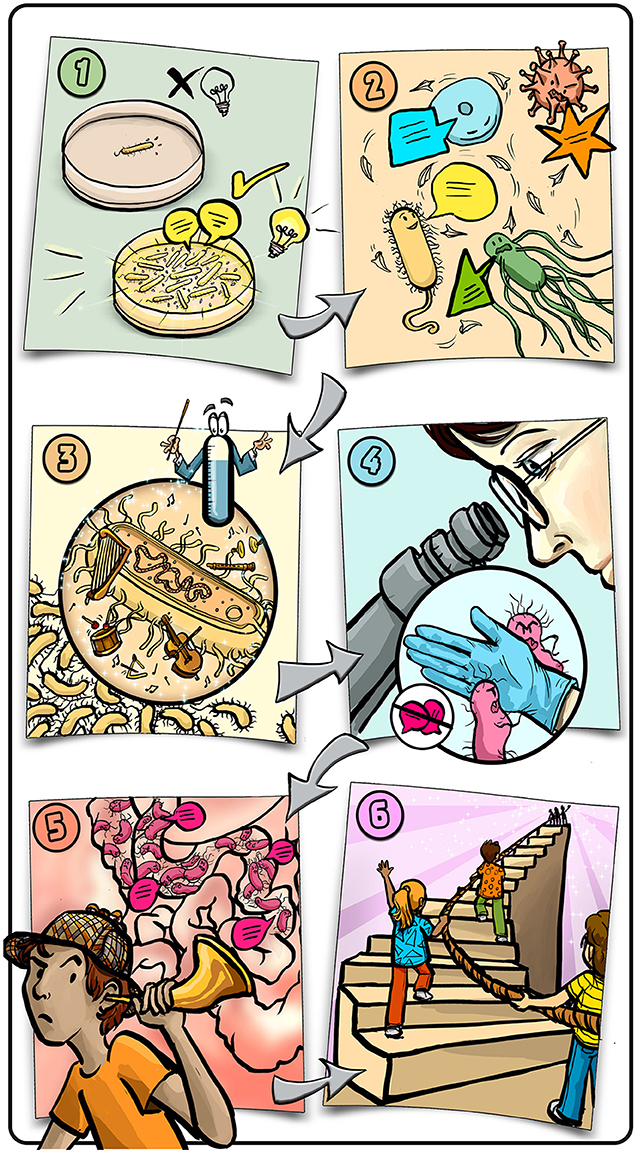
- 图文摘要 - 文章概要
- (1)研究人员发现发光细菌仅在同类聚集时才会发光, 便推测这些细菌会释放名为“自诱导物”的化学信号, 这种物质在环境中累积至一定浓度后将触发发光行为;(2)进一步研究表明细菌利用自诱导物进行交流——包括同种细菌之间、不同菌种之间、与病毒以及真核细胞的通信;(3)这种被称为“群体感应”的通信机制可协调细菌内部基因的开启与关闭;(4)科学家正在研发通过干扰有害细菌通信来治疗感染的新方法;(5)关于群体感应的知识有助于我们“监听”体内细菌的交流活动;(6)科学家历经数十年的多阶段研究才取得这一发现, 只要坚持不懈地努力, 未来的你也能做到!插图: Iris Gat.
从发光细菌中发现一种古老的语言
故事要从一种会发光的微小细菌说起。20 世纪 70 年代, Ken Nealson 与 Woody Hastings 发现, 一种名为费氏弧菌(Vibrio fischeri)的海洋发光细菌仅在同类大量聚集时才会发光 [1]。两位科学家还注意到, 当足够多的费氏弧菌存在时, 它们会同时开始发光。由此, 他们推测这些细菌会分泌一种名为自诱导物的化学物质, 当细菌密度达到临界值时, 自诱导物浓度升高至足以触发发光行为的水平。这项发现意义非凡, 它表明细菌也会彼此交流, 彻底颠覆了“细菌是‘独来独往’的简单生物”的传统观点。
现在, 我们三人(Peter、Mike 和 Bonnie)通过数十年的研究揭示了这种细菌通信的奥秘, 证实了 Ken Nealson 与 Woody Hastings 的理论(参见 专栏 1)。细菌不仅经常彼此通信, 还能与其他细胞及生物体共享关于自身和环境的复杂信息。
附表 1 - 关于细菌通信机制的主要发现
20 世纪 80 年代初, Mike 等人率先发现了费氏弧菌(Vibrio fischeri)中负责生物发光的基因(图 1A)[2]。他们的研究表明, 当这些基因被植入其他种类的细菌时, 受体细菌同样会获得发光能力!随后, Peter 将 Mike 团队发现的基因植入研究中常用的大肠杆菌(Escherichia coli), 深入探索细菌通信过程 [3], 最终证实了不同种类的细菌采用相同的通信机制。他们还发现绿脓杆菌(Pseudomonas aeruginosa)的毒力正是由这种通信机制调控, 而此种细菌会在囊性纤维化等病症患者身上引发危险的肺部感染(图 1B)。1994 年, Peter 团队受法律术语“ 法定人数”(quorum, 指重要会议所需的最低出席人数)启发, 首创了“群体感应”(quorum sensing)一词来描述这种基于化学信号的细菌通信机制。1990 年加入 Mike 实验室的 Bonnie 则对另一种发光细菌——哈维氏弧菌(Vibrio harveyi)进行研究, 发现该菌通过一种自诱导物与同种细菌交流, 还有另一种自诱导物竟是多种细菌的“通用语言” [4, 5](图 1C)。后来, Bonnie 还发现了更多类型的自诱导物, 并证实细菌不仅通过这些物质彼此交流, 还能与病毒等其他生物体通信 [6]。她的进一步研究表明, 干预群体感应可治疗动物体内的某些细菌感染 [7]。
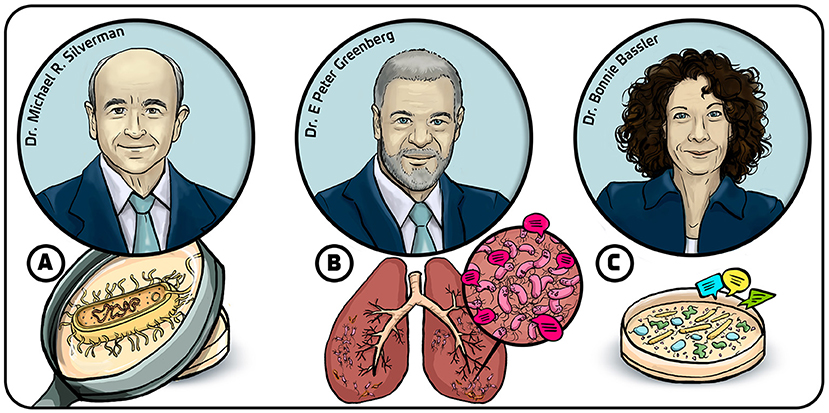
- 图 1 - 三位博士的关键发现: (A) Silverman 博士发现了费氏弧菌中负责生物发光的基因; (B) Greenberg 博士在常引发肺部感染的绿脓杆菌中识别并研究了群体感应; (C) Bassler 博士发现细菌通过多种自诱导物进行交流——不仅可与同种交流, 还能与其他菌种及病毒通信。
- 插图: Iris Gat。
群体感应——世界上最早的通信机制
作为地球上最古老的生物, 细菌拥有约 5, 000 个基因, 其中多达 600 个基因受群体感应调控。这意味着细菌基因组中近四分之一的基因就像一支由群体感应指挥的交响乐团。在交响乐团中, 指挥家不会让所有乐器同时演奏——某个时刻可能需要小提琴声部加入, 另一时刻可能需要铜管声部配合。在细菌“乐团”中也是这样: 不同浓度的自诱导物会激活特定的基因“乐器”, 当自诱导物浓度变化时, 某些基因可能会“停止演奏”并被关闭。
群体感应是地球上最早出现的通信方式, 也是最古老的社交行为。细菌个体微小难以独立成事, 但当它们通过群体感应收发周围细菌数量及亲缘关系信息时, 便能像多细胞生物体那样行动。
目前, 我们已知群体感应语言中至少存在四类自诱导物, 即“化学信号”(图 2): 第一类具有菌种特异性, 使细菌能与同种成员交流, 相当于宣告“ 你是我的同类”;第二类由遗传相近(非相同)的细菌产生, 传递“你是我的亲属”信息;第三类由多种细菌共享, 表明“我是细菌”, 用于跨菌种通信。我们认为细菌通过此类自诱导物统计所处环境中的细菌总量, 甚至可结合第一类自诱导物计算自身菌种在环境中的占比(用“同类”分子数量除以“细菌”分子数量)。最新发现的第四类自诱导物由两种分子组成, 分别用于识别“你是真核生物”和“你是病毒”。通过这四类“ 信号”, 细菌能找到同种伙伴、感知周围其他菌种, 并辨别其他类型的生物体。
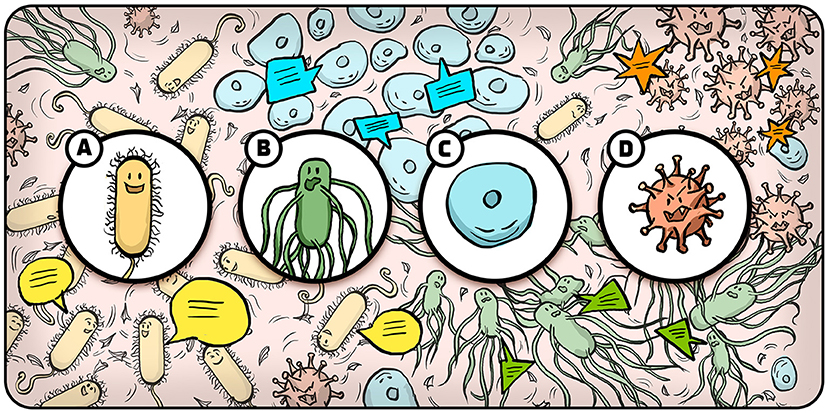
- 图 2 - 细菌的四类通信方式。
- 通过群体感应, 细菌可: (A) 与同种细菌交流; (B) 与其他种类细菌通信; (C) 与真核细胞交流; (D) 与病毒通信。插图: Iris Gat。
新型抗生素?
群体感应对人类健康具有重要意义, 因为致病菌正是利用这种机制实施攻击。致病菌携带的毒力基因全部由群体感应调控。例如, 有些基因帮助细菌形成难以清除的生物被膜, 有些则控制细菌在最佳时机释放毒素, 对宿主产生最有效的攻击。传统抗生素通过杀死细菌或抑制其繁殖来治疗感染, 但总有一部分具有抗生素耐药性的细菌能存活并不断增殖(详见本刊专文)。是否存在其他方法能消灭有害细菌?或许可以通过干扰细菌通信来降低其危害性?
科学家正在研发新型抗生素, 通过阻止细菌探测或释放自诱导物来中断其通信(图 3)。当群体感应被阻断时, 细菌便无法协调攻击行为, 由此大幅降低致病性。与传统抗生素不同, 群体感应干扰类药物不影响细菌生长也不杀死细菌, 因此科学家预计细菌对此类药物产生耐药性的速度将大幅减缓。
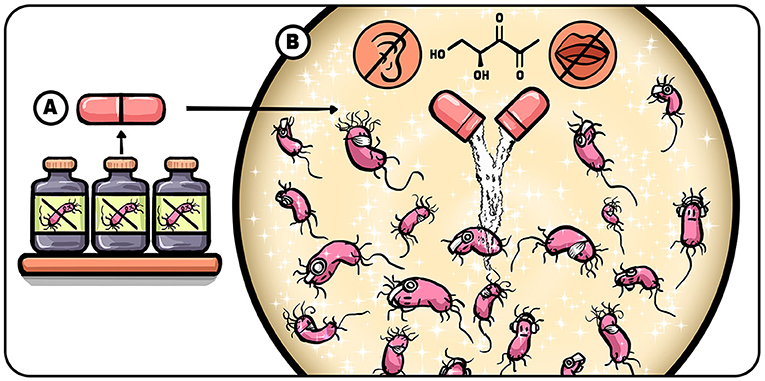
- 图 3 - 基于群体感应的新型抗生素: (A) 研究人员正基于群体感应原理开发新型抗生素; (B) 这类抗生素通过两种方式干扰细菌通信: 使细菌无法“发声”(即无法释放自诱导物), 或使其无法“聆听”其他细菌的信号(即无法感知环境中的自诱导物)。
- 插图: Iris Gat。
我们还可以用群体感应来做什么?
群体感应研究发展迅速, 我们不断发现具有迥异特性的新型群体感应分子。这些分子携带的信息可能比我们原先设想的更为复杂!一个极具吸引力的研究方向是人类微生物组内的细菌通信, 微生物组即人体内所有细菌及其他微生物的集合 [6]。人类微生物组能与人体进行交流, 对维持机体健康功能至关重要, 甚至被视为由非人类细胞构成的“新器官”。例如, 肠道微生物组与免疫系统及其他身体系统相互作用, 甚至可能影响心理健康。我们希望能“监听”肠道微生物组中细菌之间以及细菌与其他微生物的通信——就像调查人员窃听嫌疑人的电话。绘制微生物组内生物体的通信图谱, 有望为人类健康研究带来重要突破。
我们还希望利用群体感应研究细菌群落如何协同合作, 以及如何应对不守“规矩”的“作弊者”。这些作弊者不会帮着生产群落所需的必要物质, 习惯白吃白喝, 所以比合作者更具生存优势。既然作弊行为如此有利可图, 为何作弊者没有占领整个种群?研究发现, 作弊者不会激活群体感应控制的基因, 这不仅使它们不生产必需物质, 还使其易受群体中释放的毒素的影响, 而激活群体感应基因的合作者会启动抗毒素基因, 所以受毒素影响很小。这就是细菌种群维持合作的机制, 我们认为可以利用这种分子层面的认知来理解自然界中其他类型的合作行为。
热爱大自然
我们三人都对大自然怀有深切的热爱, 并选择通过科学来表达这种情感。实际上, 研究与亲近自然的道路多种多样, 每一条道路都能带来满足与喜悦。你们中有人可能想成为医生, 有人可能渴望深入丛林, 观察珍奇异兽。无论选择哪条道路, 只要能接触自然的美与奥秘, 便是值得追寻的精彩旅程。
若选择科学之路, 不妨将其视作一场寻宝之旅。那些“宝藏”——即茅塞顿开的时刻和取得的科学发现——固然极其重要且令人振奋, 但它们不会经常出现。在漫长的工作周期中, 我们时常久久不见成果, 此时必须想方设法保持好奇心与热情, 不断追寻。即便发现宝藏后, 往往也需要时间让其他科学家——甚至发现者本人——充分理解其价值。群体感应的研究正是如此, 任何新开拓的科学领域都需要时间来积累足够的数据才能产生影响力。所幸, 我们拥有众多同样热爱自然的同事和学生, 这让整个探索过程充满乐趣。
年轻人常认为自己无法取得我们这样的成就, 其实几十年前我们都是和你们差不多的学生!成为优秀科学家需要时间积累。我们相信, 只要像我们一样孜孜不倦, 任何学生都能有所成就。以杰出科学家为终极榜样固然有其价值, 但我们更建议在职业生涯的每个阶段选择与当前阶段更接近的榜样, 他们能成为助你抵达终极目标的阶梯。
术语表
生物发光 (Bioluminescent): ↑ 生物体产生并发射光的现象。
自诱导物 (Autoinducer): ↑ 一种用于细菌通信的化学物质, 帮助细菌感知环境中其他细菌及生物体的数量。
毒力 (Virulence): ↑ 细菌(及其他微生物)对其感染宿主的损伤能力。
群体感应 (Quorum Sensing): ↑ 细菌通过名为自诱导物的化学物质进行通信的机制。群体感应调控着细菌的群体行为, 如生物发光和毒力。
生物被膜 (Biofilms): ↑ 细菌彼此黏结并附着于肠道内壁等各种表面形成的菌落聚集体。
抗生素耐药性 (Antibiotic Resistance): ↑ 细菌对特定抗生素不敏感且在此类药物环境中仍能继续繁殖的特性。
微生物组 (Microbiome): ↑ 特定环境(如人类肠道)中存在的所有微生物集合。
利益冲突声明
作者声明本研究不涉及任何潜在商业或财务关系。
补充资料
致谢
感谢 Noa Segev 为本研究进行访谈并参与撰文, 同时感谢 Iris Gat 提供所有插图。
AI 人工智能工具使用声明
本文中所有图表附带的替代文本(alt text)均由 Frontiers 出版社在人工智能支持下生成。我们已采取合理措施确保其准确性,包括在可行情况下经由作者审核。如发现任何问题,请随时联系我们。
参考文献
[1] ↑ Nealson, K. H., Platt, T., and Hastings, J. W. 1970. Cellular control of the synthesis and activity of the bacterial luminescent system. J. Bacteriol. 104:313–22. doi: 10.1128/jb.104.1.313-322.1970
[2] ↑ Engebrecht, J., Nealson, K., and Silverman, M. 1983. Bacterial bioluminescence: isolation and genetic analysis of functions from Vibrio fischeri. Cell. 32:773–81. doi: 10.1016/0092-8674(83)90063-6
[3] ↑ Fuqua, W. C., Winans, S. C., and Greenberg, E. P. 1994. Quorum sensing in bacteria: the LuxR-LuxI family of cell density-responsive transcriptional regulators. J. Bacteriol. 176:269–75. doi: 10.1128/jb.176.2.269-275.1994
[4] ↑ Bassler, B. L., Wright, M., Showalter, R. E., and Silverman, M. R. 1993. Intercellular signalling in Vibrio harveyi: sequence and function of genes regulating expression of luminescence. Mol. Microbiol. 9:773–86. doi: 10.1111/j.1365-2958.1993.tb01737.x
[5] ↑ Chen, X., Schauder, S., Potier, N., Van Dorsselaer, A., Pelczer, I., Bassler, B. L., et al. 2002. Structural identification of a bacterial quorum-sensing signal containing boron. Nature. 415:545–9. doi: 10.1038/415545a
[6] ↑ Duddy, O. P., and Bassler, B. L. 2021. Quorum sensing across bacterial and viral domains. PLoS Pathog. 17:e1009074. doi: 10.1371/journal.ppat.1009074
[7] ↑ Papenfort, K., and Bassler, B. L. 2016. Quorum sensing signal–response systems in Gram-negative bacteria. Nat. Rev. Microbiol. 14:576–88. doi: 10.1038/nrmicro.2016.89
