摘要
细菌是一种存在于微观世界的单细胞生物, 几乎无处不在, 不仅存在于我们周围的环境中, 也生活在我们自身体内。细菌的种类非常多, 它们的形状、大小各不相同, 生长和生活方式也有着显著的差异。有的细菌能够迅速生长, 而有的则生长较慢。我们非常想了解环境中各种不同类型细菌的生长情况, 但遗憾的是, 有些细菌在实验室里很难培养。为了克服这一难题, 科学家们发明了一种新方法, 通过分析细菌的 DNA, 就能预测出它们的生长速度。这意味着, 即便我们无法在实验室中培养某些细菌, 我们也能通过它们的 DNA 了解它们的生长速度。
有些细菌生长迅速, 有些则不然
我们的地球上大部分生命都是微小的存在, 其中细菌就是典型的单细胞微生物, 它们在微观世界中生活。不管是在哪里, 包括我们自己的身体内, 都能找到细菌的踪迹。更令人惊奇的是, 细菌的种类繁多, 形态和大小千差万别。它们在生长和生活方式上也表现出极大的多样性。比如, 有的细菌可以迅速繁殖, 而另一些则生长得非常缓慢, 有的甚至可能需要数千甚至数万年才能完成一次繁殖 [1]。作为科学家, 我们对于为什么有些细菌生长迅速, 而其他一些却生长缓慢这一现象充满好奇。例如, 如果一种细菌既能导致疾病, 又能快速繁殖, 那它可能会成为一个十分危险的存在。
细菌的繁殖过程被称为二分裂。在这个过程中, 一个单细胞会生长并复制它的 DNA, 最后分裂成两个“子”细胞 (如图 1 所示)。这就像细菌在进行自我克隆, 制造出一个与自己几乎完全相同的副本。不同细菌的分裂速度差异很大。有些快速生长的细菌能在短短 10 分钟内完成一次分裂。而另一些生长缓慢的细菌可能需要几天甚至更长时间才能分裂一次。例如, 肠道沙门氏菌 (Salmonella enterica), 这种在肠道中生长可能导致食物中毒的细菌, 大约每 20 分钟就能分裂一次;而原绿球藻 (Prochlorococcus), 作为世界海洋中数量最多的生物之一 [2], 可能需要一天或更长的时间才能完成一次分裂。我们用“倍增时间”这个术语来描述细菌细胞分裂所需的时间(见图 1)。通过观察它们的倍增时间, 我们能够测量细菌种群的生长速度。细菌快速生长意味着它们的倍增时间较短, 而生长缓慢的细菌则有较长的倍增时间。
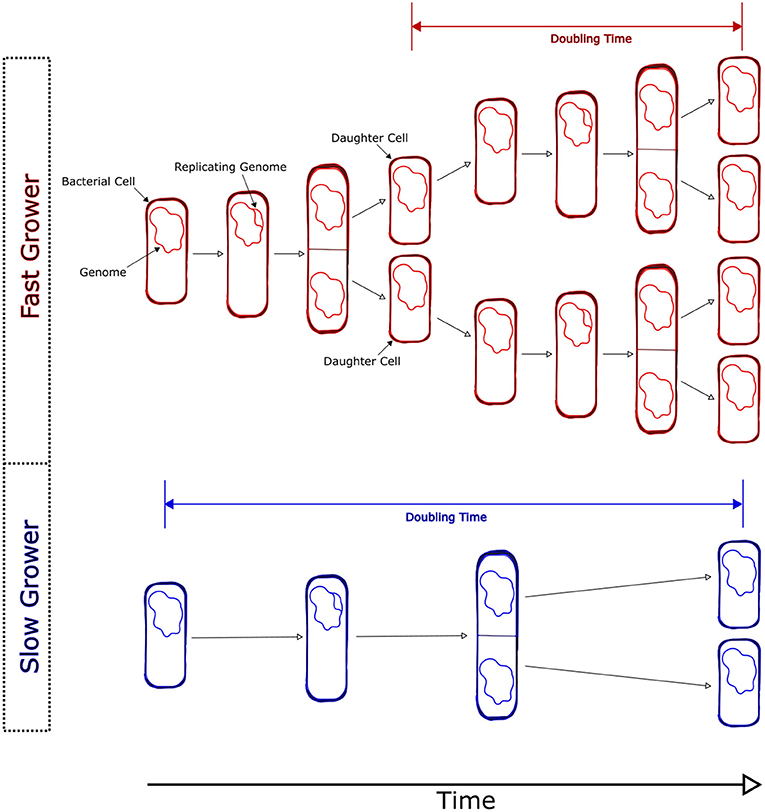
- 图 1 - 细菌的繁殖过程被称为二分裂。
- 在这个过程中, 随着细胞的生长, 它们会变大, 并开始复制自己的基因组 (也就是 DNA)。最后, 一个细菌细胞会分裂成两个新的细胞, 每个“ 子”细胞都会获得一份基因组的副本。这个从一个细胞分裂为两个细胞的时间间隔就被称为倍增时间。生长速度快的细菌拥有较短的倍增时间, 而那些生长较慢的细菌则有较长的倍增时间。
细菌并非在所有环境下都能快速生长——它们需要合适的条件。例如, 当食物充足时, 细菌会快速生长, 倍增时间较短。但当资源变得稀缺时, 它们的分裂速度可能会减慢, 导致倍增时间变长。这就是为什么我们要小心食物变质——对于微小的细菌来说, 即使是少量的人类食物也可能是丰富的食源, 因此它们会开始迅速分裂。而另一方面, 我们通常不太担心家中的大多数表面会积聚大量细菌, 因为那些地方通常没有太多适合细菌生长的食物。尽管舔墙壁听起来很恶心, 但这种行为可能不会像食用变质食物那样导致你生病 (不过在读完这篇文章之后, 也请不要去舔墙壁哦!你永远不知道那上面会有什么……)。
食物的数量并不是影响细菌生长的唯一因素。有些细菌本身就比其他细菌生长得更快。也就是说, 不同种类的细菌有着不同的最短倍增时间。最短倍增时间是指在理想条件下细菌数量翻倍所需的最短时间。也就是说, 即使细菌有充足的食物, 它们也不一定能够快速生长。这就像是, 如果你又饿又渴, 可能无法像吃饱喝足时跑得那么快。但无论你吃得多饱, 也不可能跑得像猎豹那样快, 毕竟, 在进化过程中, 有些物种本身就具备比其他物种跑得更快、或生长得更快的能力。
如何在不培养某微生物的情况下测量其生长速度?
遗憾的是, 我们还无法精确测量许多细菌的生长速度, 原因是这些细菌在实验室里难以培养。要弄清楚一个细菌种类的理想生长条件——比如它喜欢什么类型的食物、它适宜的生长温度, 以及合适的盐度和 pH 值——这可不是件简单的事。这个过程不仅成本高, 而且时间长。尽管科学家们估计地球上有大约 40 万种细菌 [3], 但我们目前只了解其中大约 1.4 万种的生长条件。那些我们在实验室里无法培养的细菌仿佛是一道未解之谜。而要解开这个谜团, 从而探索它们在自然界中的角色, 了解它们的生长速度是关键的第一步。
那么, 科学家们对此能做些什么呢?实际上, 即使我们无法在实验室中培养某种细菌, 我们仍然能够获取许多关于它的信息。那么, 这是如何实现的呢?我们可以直接从这些细菌的自然生存环境中收集细菌样本, 并对它们的 DNA 进行检测, 而不需要将它们培养出来。这里我们不深入细节, 但关键是, 即便我们无法在实验室中培养这些细菌, 有时我们还是能够恢复它们的 DNA 序列。通过分析细菌的 DNA 序列, 我们可以了解到该细菌种类的生长速度有多快。
细菌基因组中的生长线索
那我们怎样才能仅通过细菌的 DNA 序列来推断它们的分裂速度呢?在解释如何从 DNA 预测生长速度之前, 我们需要先简单了解一下 DNA 是怎样发挥作用的。想象一下, 每个生物都是由许多细胞组成的, 而每个细胞内都含有 DNA, 这些 DNA 就像是一个建造生物的“说明书”——它们提供了细胞生存和生长所必需的所有指令。我们把细胞内所有的 DNA 集合称为基因组。基因是一段段的 DNA, 每一段 DNA 都负责告诉细胞如何制造特定的蛋白质, 而蛋白质则构成细胞机器的分子。
这的确是一大堆信息需要消化!让我们用一个更简单的方式来理解它。想象基因组就像是一本构建细胞的说明手册, 而每个基因就好比手册里的一个“章节”, 它包含了构建一个细胞的具体指令(参见图 2A)。那么, 如果把基因比作说明手册的章节, 那组成各个章节的单词又是什么呢?DNA 由四个字母组成: A、T、G 和 C。每一个字母代表一种不同类型的分子, 这些分子可以连接成更长的 DNA 链。所有的遗传“单词”都是由三个字母组成, 我们称它们为密码子。每一个单词编码一个氨基酸, 氨基酸是蛋白质的基本构成单元。然后, 一连串的词 (即基因或“章节”) 组合起来, 用于构建在细胞中发挥特定功能的特定蛋白质。
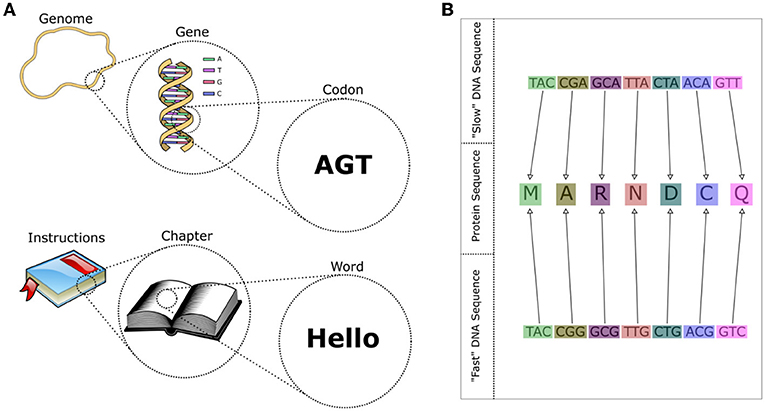
- 图 2 - (A) 可以把基因组想象成构建细胞的一本“说明手册”, 其中每个基因都是手册中的一个“章节”, 而每个密码子则是一个“单词”。
- 所有密码子都由三个字母组成——在“DNA 语言”中, 每个单词都只有三个字母 (见图 B)。DNA 语言具有冗余性, 这意味着多个不同的“单词” (即密码子) 可以表达相同的含义。因此, 相同的蛋白质序列可以由不同的 DNA 序列生成。但某些序列比其他序列更容易被细胞快速生成, 从而使细胞生长得更快。
就像英语或任何其他语言一样——不同的“单词”可以表达相同的意思—— DNA 的语言也有其冗余性, 不同的密码子可以编码相同的氨基酸, 即蛋白质的一部分 (参见图 2B)。而且, 正如人们在使用语言时有所偏好一样, 细胞在选择密码子“单词”时也有其偏好。比如, 大多数人更倾向于说 “我饿了”, 而不是用更复杂的表达“我饥饿”。尽管这两种表达字数相同, 但对大多数人来说, 最先想到的往往是“饿了”, 而不是“饥饿”。也就是说, 即使我们懂得某些复杂词汇的含义, 我们也可能不喜欢使用它们, 追求沟通效率的时候尤其如此。同理, 虽然所有细菌种类的“词汇表”中都包含相同的密码子, 但每种细菌对密码子的偏好各不相同。当细胞需要快速生长时, 它们更倾向于使用自己偏好的密码子来构建蛋白质。随着时间的推移, 在自然选择的作用下, “词汇量”较小的细胞种类便能生长得更快。因此, 通过测量某种细胞的偏好词汇量的大小, 我们就可以预测它的生长速度——偏好词汇量越小, 生长速度越快, 倍增时间越短。
我们开发了一个计算机模型 [4], 测量某种细菌的密码子“词汇表”大小, 以此来预测这些细菌的生长速度 (见图 3)。这样一来, 即使我们不能在实验室中培养某些细菌, 只要我们已经测序了该细菌种群的基因组, 我们就能估计它们的生长速度。这意味着, 我们现在可以测量更多种类细菌的生长速度, 大大超越了以前的能力。
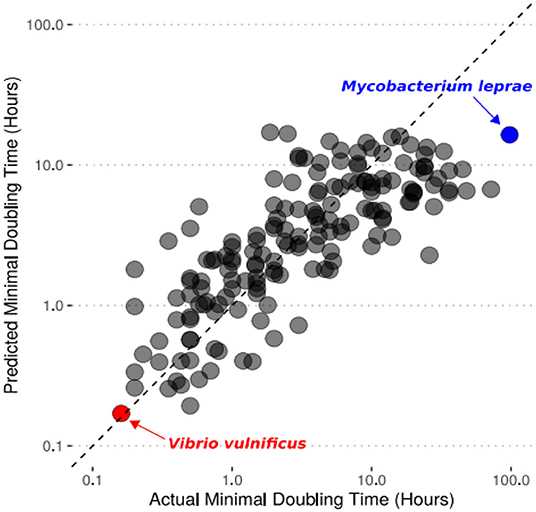
- 图 3 - 我们的计算机模型能够预测细菌物种的倍增时间, 这些预测与实验室中实际测量得到的最小倍增时间非常接近。
- 在图 3 中, 虚线代表的是完美预测的情况, 也就是说预测值和实际值完全一致。图表上的每个点都代表我们尝试预测生长速度的不同细菌。我们特别用红色标出了创伤弧菌 (Vibrio vulnificus), 因为它是我们预测中生长速度最快的细菌。同时, 我们还用蓝色标出了麻风分枝杆菌 (Mycobacterium leprae), 预测中生长速度最慢的细菌。
培养生长缓慢的细菌很困难
我们使用计算机模型发现, 我们在实验室里能够培养的细菌大多数都是生长速度非常快的。想象一下, 如果你是一位科学家, 你会愿意花很长时间等待一种细菌慢慢生长吗?更麻烦的是, 很多生长缓慢的细菌还特别挑剔, 要确定它们理想的生长条件并不容易。总的来说, 在实验室中培养生长缓慢的细菌既耗时又枯燥。这就意味着, 相比那些生长迅速的细菌, 我们对生长缓慢的细菌其实知之甚少。
当我们预测从自然环境中直接采集的细菌的倍增时间时, 我们发现这些细菌的倍增时间通常比我们在实验室中能够培养的细菌的倍增时间要长得多。这解释了为什么过去科学家们更多地研究那些生长速度快的细菌。科学家们的时间和资源都是有限的, 他们有时倾向于选择那些最容易培养的细菌进行研究, 而不是那些在自然环境中更常见的细菌。然而, 现在有一些科学家正致力于了解更多关于生长缓慢的细菌的知识。虽然培养这些细菌在未来仍将是一个挑战, 但关于它们的生物学还有很多未解之谜, 等待我们去探索, 认识这些细菌是非常重要的, 因为在某些环境中, 比如海洋, 其中的大部分细菌都是生长缓慢的细菌。
前瞻性思考
DNA 能揭示关于细菌生活方式的许多信息。在这篇文章中, 我们主要探讨了 DNA 如何帮助我们了解细菌的生长速度。然而, 除此之外, DNA 还隐藏着许多关于细菌的其他有趣的秘密。那么, DNA 还能告诉我们哪些关于细菌的信息呢?我们还可以研究哪些细菌的特性?想象一下, 如果你能使用 DNA 来预测关于细菌的某些特性, 你最感兴趣的是什么?
研究资金支持
JW 获得了西蒙斯基金会提供的 653212 号海洋微生物生态学博士后奖学金。我们还要感谢西蒙斯基金会海洋生态系统计算生物地球化学建模合作项目 (CBIOMES, 项目编号 549943) 和美国国家科学基金会海洋科学部 (OCE, 项目编号 1737409) 对 JF 的资金支持。
术语表
二分裂 (Binary Fission): ↑ 细菌的繁殖过程, 在这个过程中, 一个细胞分裂成两个相同的“子”细胞。
倍增时间 Doubling Time: ↑ 细胞通过分裂实现数量翻倍所需的时间。生长速度快的细菌倍增时间较短, 生长速度慢的细菌的倍增时间会更长一些。
基因组 Genome: ↑ 细胞的所有 DNA, 细胞的“说明书”。
基因 Gene: ↑ 提供构建蛋白质指令的 DNA 片段。
蛋白质 Proteins: ↑ 发挥生命功能的细胞的重要组成部分。
密码子 Codon: ↑ 一个 DNA “单词”, 编码蛋白质的一个特定部分。
氨基酸 Amino Acid: ↑ 蛋白质的构建块。每个氨基酸由一个或多个密码子编码。
计算机模型 Computer Model: ↑ 为更好地理解和预测生物学原理而搭建的电脑模型。
利益冲突声明
作者声明, 该研究是在没有任何可能被解释为潜在利益冲突的商业或财务关系的情况下进行的。
原文
↑ Weissman, J. L., Hou, S., and Fuhrman, J. A. 2021. Estimating maximal microbial growth rates from cultures, metagenomes, and single cells via codon usage patterns. Proc. Natl. Acad. Sci. U.S.A. 118:e2016810118. doi: 10.1073/pnas.2016810118
参考文献
[1] ↑ Jørgensen, B. B., and D’Hondt, S. 2006. Ecology-A starving majority deep beneath the seafloor. Science. 314:932–4. doi: 10.1126/science.1133796
[2] ↑ Partensky, F., Hess, W. R., and Vaulot, D. 1999. Prochlorococcus, a marine photosynthetic prokaryote of global significance. Microbiol. Mol. Biol. Rev. 63:106–27. doi: 10.1128/MMBR.63.1.106-127.1999
[3] ↑ Lewis, W. H., Tahon, G., Geesink, P., Sousa, D. Z., and Ettema, T. J. 2020. Innovations to culturing the uncultured microbial majority. Nat. Rev. Microbiol. 22:1–6. doi: 10.1038/s41579-020-00458-8
[4] ↑ Weissman, J. L., Hou, S., and Fuhrman, J. A. 2021. Estimating maximal microbial growth rates from cultures, metagenomes, and single cells via codon usage patterns. Proc. Natl. Acad. Sci. U.S.A. 118:e2016810118. doi: 10.1073/pnas.2016810118