Résumé
Les protéines sont de petites machines biologiques qui travaillent dans notre corps comme chez tous les autres animaux, les plantes, les bactéries et les virus. Elles assurent un large éventail d’activités vitales pour ces organismes. Les protéines sont synthétisées sur la base d’instructions contenues dans le support de notre information génétique, notre ADN, par des organites cellulaires appelés ribosomes. Les ribosomes assemblent les protéines très rapidement et avec précision. La plupart de mes recherches scientifiques sont consacrées à la compréhension de la fonction du ribosome, basée sur sa structure, que nous avons déterminée. Dans cet article, je décris les principales conclusions de ma recherche, certaines applications importantes de ces découvertes et les défis auxquels j’ai été confrontée en cours de route.
La professeure Ada Yonath a reçu le prix Nobel de chimie en 2009, partagé avec les professeurs Venkatraman Ramakrishnan et Thomas A. Steitz, pour leurs études sur la structure et la fonction du ribosome.
De l’adn aux protéines
Chaque cellule vivante possède un matériel génétique, qui contient, sous forme d’un code, les instructions pour la fabrication des composants cellulaires de l’organisme vivant entier. Il est transmis par les parents à leur progéniture. Le matériel génétique est appelé ADN et peut être considéré comme un livre de cuisine qui contient toutes les informations nécessaires à la création et au fonctionnement de l’organisme vivant. L’un des composants les plus importants de l’ADN sont les gènes, qui contiennent les instructions pour la synthèse des protéines – de minuscules ≪ machines ≫ des cellules et des organismes vivants. Les protéines effectuent de nombreuses réactions importantes pour le fonctionnement de tous les organismes.
Toutes les protéines naturelles sont constituées d’une combinaison de 20 éléments de base appelés acides aminés. Pour former une protéine, les acides aminés sont réunis comme des perles sur une chaîne. La connexion entre deux acides aminés est appelée liaison peptidique, et une protéine de taille moyenne contient un assemblage d’environ 150 à 500 acides aminés. Chacune de ces longues chaînes d’acides aminés se replie en une structure tridimensionnelle unique et fonctionnelle déterminée par la séquence des acides aminés qui la composent. La structure protéique est précisément adaptée pour exécuter la fonction que la protéine est censée remplir (Figure 1).
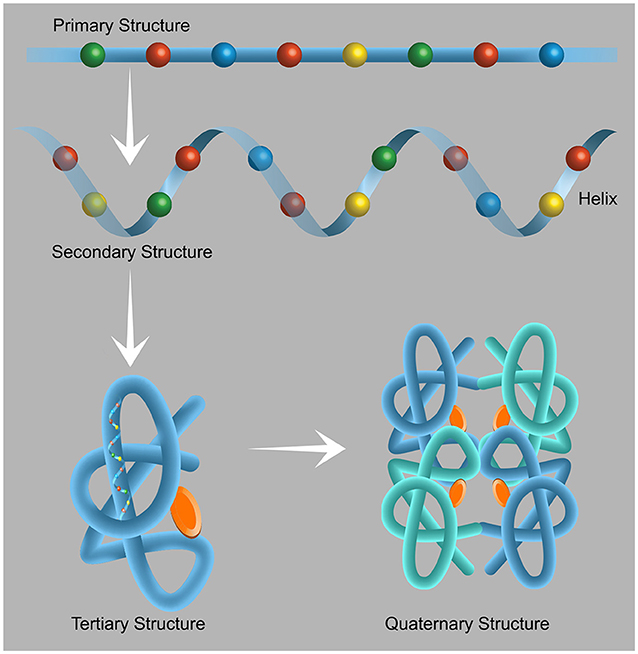
- Figure 1 - La structure des protéines.
- Les protéines sont constituées de 20 éléments de base appelés acides aminés. Lorsque les acides aminés sont réunis, ils forment une chaîne appelée structure primaire (primary structure) de la protéine. Chaque acide aminé a des propriétés chimiques uniques nécessaires au fonctionnement de la protéine. L’ordre dans lequel les acides aminés sont assemblés est extrêmement important. Chacune des chaînes protéiques se replie selon les propriétés de ses acides aminés pour former la structure secondaire (secondary structure), qui peut prendre diverses formes, dont celle d’une hélice. La structure tertiaire (tertiary structure) est la structure tridimensionnelle de la protéine, c’est-à-dire l’arrangement des hélices et des chaînes reliant les hélices qui permet l’exécution des fonctions spécifiques de cette protéine. Certaines protéines sont actives sous forme d’assemblages – leur arrangement est alors appelé structure quaternaire (quaternary structure). Ces assemblages sont constitués d’au moins deux composants protéiques liés ensemble de manière à permettre leur activité. Figure adaptée de https://biologydictionary.net/protein-structure/.
La production de protéines dans les cellules se fait en plusieurs étapes (Figure 2). Dans la première étape, une partie de l’ADN, est copiée en ARN messager (ARNm) par un processus appelé transcription. Une molécule d’ARNm est une ≪ copie ≫ de l’information génétique de l’ADN qui permet de transférer cette information à l’intérieur de la cellule sans ≪ mettre en danger ≫ l’information génétique originale. Dans l’étape suivante, l’ARNm atteint le ribosome, ≪ l’usine ≫ de la cellule pour la production de protéines. Le ribosome ≪ lit ≫ les instructions de l’ARNm et fabrique la protéine désirée en formant une chaîne d’acides aminés reliés les uns aux autres par des liaisons peptidiques. Après être sorties du ribosome, les chaînes protéiques se replient spontanément dans la structure tridimensionnelle appropriée nécessaire au fonctionnement de la protéine. Ce processus de production de protéines dans le ribosome est appelé traduction. Le reste de cet article se concentrera sur la manière dont fonctionne le processus de traduction dans le ribosome.
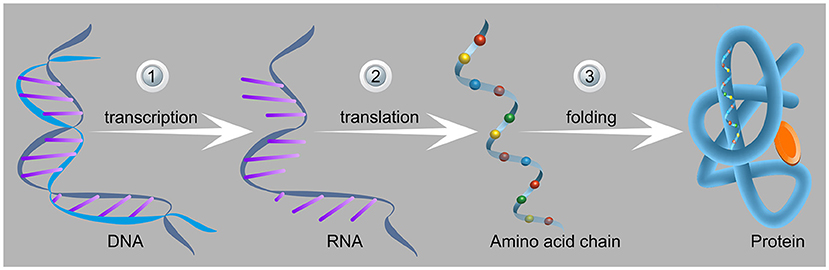
- Figure 2 - Étapes de la production de protéines dans les cellules.
- (1) Dans la première étape, le gène, partie de l’ADN (DNA) contenant l’enchaînement de nucléotides (séquence) correspondant à une protéine (protein) (à gauche), est transcrit en ARN messager. (RNA) (2) Dans la deuxième étape, appelée traduction (translation), l’ARN messager atteint le ribosome où est fabriquée la chaîne d’acides aminés (amino acid chain) (à savoir la protéine) qui quitte le ribosome par un tunnel protégé. (3) Après être sortie du ribosome, la chaîne se replie (folding) dans la structure tridimensionnelle de la protéine (à droite). Cette structure unique est appropriée à la fonction de la protéine (l’une des nombreuses fonctions différentes des différentes protéines dans l’organisme). Image adaptée de : http://bio.academany.org/2017/labs/BioRiiDL_2017/sreejith/week2.html.
Le ribosome
La traduction d’une nouvelle protéine est un processus compliqué. La ≪ recette ≫ des protéines est inscrite dans le code génétique (ADN et ARNm), dans un ≪ langage ≫ génétique de quatre lettres qui est traduit en un ≪ langage ≫ d’acides aminés contenant 20 éléments de base différents : les acides aminés. Tu te demandes probablement : ≪ Comment est-ce possible ? ≫ Le processus est appelé traduction, et ce nom suggère le processus. La solution est que le ribosome ≪ lit ≫ le code génétique comme une série de combinaisons de nucléotides de trois lettres appelées codons. Chaque codon correspond à un acide aminé particulier (certains acides aminés ont plus d’un codon. Parfois, il y a jusqu’à six codons différents pour le même acide aminé). Trois codons ne correspondent à aucun acide aminé et constituent des signaux d’arrêt de la traduction. Le ribosome est responsable de cette activité, c’est-à-dire qu’il permet les connexions entre les acides aminés de la protéine naissante.
Le ribosome est un organite cellulaire composé de deux sous-unités : la petite sous-unité ribosomique et la grande sous-unité ribosomique (Figure 3), chacune ayant une fonction spécifique. La petite sous-unité est responsable de la lecture du code génétique de l’ARNm et des correspondances entre le codon d’ARNm et l’anticodon correspondant (complémentaire du codon) porté par un ARN de transfert (ARNt) chargé de l’acide aminé correspondant à ce codon. Le site de lecture du code génétique est appelé le centre de décodage. La grande sous-unité ribosomique sert à catalyser la nouvelle liaison peptidique entre les acides aminés et à faire passer la protéine synthétisée à travers le ribosome dans le tunnel de sortie du polypeptide naissant (NPET). Dans le centre catalytique du ribosome, grâce à son activité peptidyl transférase, les acides aminés (portés par des ARNt) se lient les uns aux autres, dans l’ordre donné par la séquence, par la formation d’une nouvelle liaison peptidique. Le centre catalytique et son activité peptidyl transférase (PTC) permettent la liaison des deux acides aminés car à l’intérieur de celui-ci, ils sont positionnés très proches et dans la bonne orientation. Une fois la nouvelle liaison formée, l’ARNt contenant la protéine naissante se déplace à travers le ribosome avec l’ARNm vers la ≪ station suivante ≫ ; un nouvel ARNt peut ainsi se positionner au PTC et une nouvelle liaison peptidique être formée. La chaîne protéique naissante traverse la grande sous-unité ribosomique au sein du NPET et émerge à l’autre extrémité du ribosome.
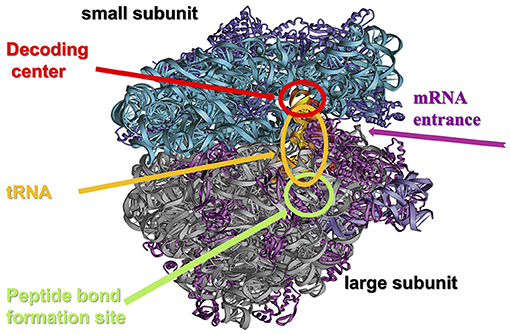
- Figure 3 - Structure du ribosome.
- Le ribosome est constitué de quelques dizaines de protéines (violet et magenta) et de chaînes d’ARN ribosomique (turquoise et gris). Il est constitué de deux sous-unités : la petite sous-unité ribosomique (small subunit) (turquoise et violet) et la grande sous-unité ribosomique (large subunit) (magenta et gris). La petite sous-unité contient le site où le matériel génétique est lu, appelé centre de décodage (decoding center), où la molécule d’ARNm (mRNA entrance) rentre et se lie à un ARN de transfert (ARNt) (tRNA) selon le code génétique, représenté par trois lettres (voir Figure 4). La grande sous-unité ribosomique contient un site appelé centre peptidyl transférase (PTC) (peptide bond formation site), où les acides aminés, transportés par l’ARNt, se lient les uns aux autres. Les deux sous-unités du ribosome s’associent au début du processus et se séparent à la fin de l’assemblage de la protéine (Crédit Figure : Ada Yonath).
Une petite et une grande sous-unité ribosomique sont associées pendant la synthèse d’une protéine et se détachent l’une de l’autre lorsque la traduction de la protéine est terminée. Chaque sous-unité ribosomique est constituée de protéines et de très longues chaînes d’ARN appelées ARN ribosomiques (ARNr) (Figure 3) [1]. Il y a beaucoup de ribosomes dans chaque cellule vivante – des dizaines de milliers dans les bactéries, des millions dans les cellules humaines. Le travail des ribosomes consomme beaucoup d’énergie, plus de 60% de l’énergie totale utilisée par la cellule [2] ! Cela souligne l’importance du ribosome dans la cellule, car c’est l’usine qui produit les protéines nécessaires à la plupart des activités cellulaires. Le ribosome fonctionne à une vitesse étonnante : un ribosome bactérien peut produire uneprotéine de taille moyenne en moins d’une minute, parfois en quelques secondes.
La traduction d’une molécule d’ARNm se produit en trois étapes : démarrage, élongation et terminaison. Pendant le démarrage, un ARNm se lie à la petite sous-unité ribosomique. Ensuite, un ARNt transportant l’acide aminé méthionine se lie au codon de départ de la séquence d’ARNm. Le codon de départ dans toutes les molécules d’ARNm a la séquence AUG, qui correspond à la méthionine. Des facteurs de démarrage rejoignent la petite sous-unité ribosomique ; avec l’ARNm et les ARNt, ils forment le complexe de démarrage. Ensuite, la grande sous-unité ribosomique se lie et forme le complexe de traduction, c’est-à-dire le ribosome actif. Pendant la phase d’élongation, le ribosome traduit chaque codon à tour de rôle. Pour chaque acide aminé, il existe un ARNt distinct et unique qui se lie au ribosome et transporte avec lui l’acide aminé. Au fur et à mesure que le ribosome se déplace à travers l’ARNm, de nouvelles molécules d’ARNt chargées de l’acide aminé correspondant continuent de se lier et de se déplacer entre les trois sites actifs du ribosome : le site Aminoacyl (site A), le site Peptidyl (site P) et le site de sortie (site E) (Figure 4). A chaque cycle, l’ARNt suivant transporte un acide aminé, pénètre dans le ribosome, se lie au site A, une liaison peptidique se forme entre les acides aminés liés au site A et au site P, et la chaîne protéique naissante se déplace dans le tunnel de sortie peptidique naissant (NPET). Ensuite, l’ARNt du site P se déplace vers le site E et est libéré du ribosome. L’ARNt du site A se déplace vers le site P, et un nouvel ARNt se lie au site A. Chaque fois qu’un nouvel acide aminé arrive, il se lie à l’acide aminé précédent par une liaison peptidique. De cette façon, la chaîne protéique naissante est formée [3]. La chaîne peptidique en croissance traverse le ribosome par le tunnel de sortie peptidique naissant (NPET) et émerge du ribosome. L’allongement se poursuit tant que l’ARNm avance dans le ribosome et présente de nouveaux codons. La terminaison se produit lorsqu’un codon d’arrêt se présente. Ce codon stop est un codon sur l’ARNm qui n’est reconnu par aucune molécule d’ARNt. Lorsque la traduction est terminée, les deux sous-unités du ribosome se séparent. La protéine naissante sort et se replie dans sa structure tridimensionnelle, seule ou avec des protéines assistantes (appelées chaperons). De cette façon, la nouvelle protéine est libérée du ribosome et peut remplir sa fonction dans la cellule. (vidéo décrivant ce processus ici : https://www.youtube.com/watch?v=Jml8CFBWcDs).
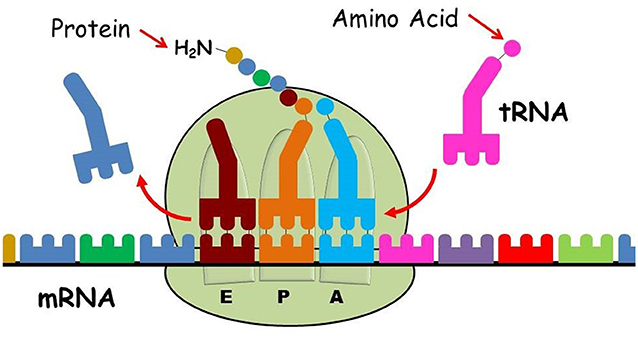
- Figure 4 - Production de protéines dans le ribosome.
- Le ribosome est composé de deux sous-unités distinctes et séparées : la petite sous-unité (en bas) et la grande sous-unité (en haut). La petite sous-unité décode l’ARNm (mRNA) (chaîne multicolore) qui contient les instructions de séquence de la protéine. La grande sous-unité catalyse la formation de liaisons peptidiques et synthétise la chaîne protéique naissante selon les instructions de l’ARNm comme suit : le premier ARN de transfert (tRNA) chargé de l’acide aminé (amino acid) correspondant (cercle coloré) se lie au site peptidyle (site P), puis l’ARNt chargé suivant se lie au site aminoacyle (site A) du ribosome. Une nouvelle liaison peptidique se forme entre les acides aminés liés à l’ARNt au site peptidylique (site P) et au site A (cercles bleu pâle et orange dans les sites A et P). L’ARNt du site P est transféré au site de sortie (site E) (rouge foncé), l’ARNt du site A est transféré au site P avec la chaîne protéique naissante qui lui est liée, et un nouvel ARNt chargé avec l’acide aminé suivant peut se lier au site A. À la fin du processus, la chaîne d’acides aminés est libérée du ribosome et se replie en une protéine fonctionnelle (Crédit image : figure Google modifiée d’Ada Yonath).
La cristallographie inspirée par les ours polaires
Nous avons élucidé la structure et la fonction du ribosome en utilisant une méthode appelée cristallographie. Cette méthode nous permet d’étudier les matériaux cristallisés. Un cristal est une structure composée d’unités à symétrie interne qui se répètent dans l’espace de manière ordonnée. L’unité répétitive la plus simple et la plus petite dans un cristal est appelée ≪ cellule unitaire ≫. Une cellule unitaire de sel de table est représentée à la Figure 5A. Chaque cellule unitaire d’un cristal a une longueur, une largeur et une hauteur fixes, ainsi que les angles entre ses faces. Dans un cristal, les cellules unitaires sont disposées périodiquement en trois dimensions, côte à côte, comme des carreaux sur un sol. En cristallographie, le cristal est irradié par un faisceau de rayons X uniforme et très concentré (Figure 5B). Les atomes présentés dans chaque cellule unitaire du cristal diffractent le faisceau de rayons X dans quelques directions spécifiques, déterminées par une loi physique, appelée loi de Bragg et selon leurs propriétés et leurs positions. Pendant l’irradiation, les images de la diffraction du faisceau sont projetées sur un écran (Figure 5B, écran à droite) ou sur un détecteur. En traitant ces images de diffraction par des outils mathématiques appelés équations de Fourier, nous pouvons déduire la structure tridimensionnelle de la cellule unitaire.
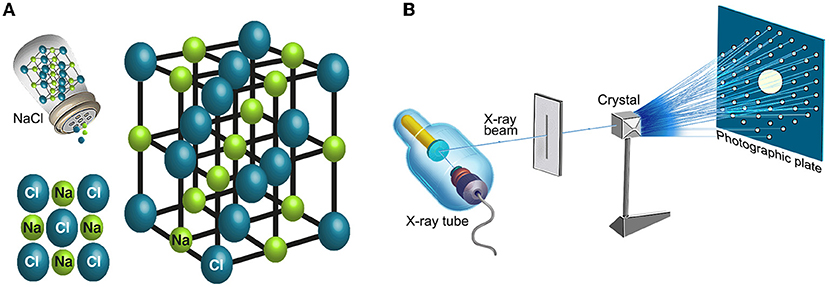
- Figure 5 - Déchiffrer la structure d’un cristal à l’aide de la cristallographie.
- (A) Un exemple d’arrangement atomique dans un cristal (crystal) connu de notre vie quotidienne – le sel de table. Le sel de table (NaCl) se compose de deux ions : le cation sodium (Na : ion positif – en vert) et l’anion chlorure (Cl : ion négatif – en bleu), disposés périodiquem- ent en cellules unitaires qui se répètent dans toutes les directions de l’espace. (B) Détermination de la structure d’un cristal par cristallographie : le cristal est irradié avec un faisceau de rayons X (X-ray tube) uniforme et concentré. En raison de la disposition des atomes qui composent le cristal, le faisceau de rayons X est diffracté après avoir frappé le cristal. Au cours de l’expérience, le cristal est irradié sous plusieurs angles, et l’intensité et la position des points de l’image projetées sur la plaque photographique (photographic plate) sont mesurées (à droite). En utilisant l’image du faisceau diffracté, nous pouvons déchiffrer la structure tridimensionnelle du cristal.
La première exigence pour déchiffrer la structure à l’aide de rayons X est la préparation de cristaux. Certains matériaux, comme le sucre, le sel de table ou le quartz, sont des cristaux naturels – ces cristaux sont stables et solides. Mais la plupart des matériaux et des structures dans la nature et dans le corps, tels que les protéines (par exemple, l’hémoglobine) ou leurs complexes, comme les ribosomes, ne forment pas naturellement de cristaux. Par conséquent, le premier défi de notre recherche a été de trouver les conditions pour cristalliser les ribosomes, à composants multiples, compliqués et flexibles – une tâche auparavant considérée comme impossible, car de nombreux scientifiques célèbres avaient déjà essayé et échoué. Pendant longtemps, nous n’avons pas pu cristalliser les ribosomes en raison de leur structure extrêmement compliquée. En raison de leurs exigences fonctionnelles dans chaque organisme, ils ont des propriétés flexibles et ont tendance à se désassembler à l’extérieur de la cellule, ce qui rend leur cristallisation encore plus difficile.
L’une des nombreuses percées importantes que nous avons faites en essayant de cristalliser le ribosome a été inspirée par les ours polaires. Il s’avère que tout au long de l’hiver, une grande partie des ribosomes des ours sont maintenus dans une structure dense, presque périodique, jusqu’à ce que les ours reprennent toute leur activité au printemps. Nous avons supposé que cela se produisait pour que les ribosomes des ours ne se désagrègent pas lorsqu’ils sont quasiment inactifs pendant l’hiver, et pour qu’ils puissent encore fonctionner au printemps. Cela nous a indiqué que, dans des conditions de stress ou extrêmes, les ribosomes peuvent avoir tendance à s’organiser dans une densité presque périodique, et nous avons donc choisi de produire des cristaux à partir de ribosomes de bactéries vivant dans des conditions de stress extrêmes dans la mer Morte. Et en effet, grâce à un travail intense et après avoir essayé d’innombrables conditions différentes, nous avons réussi à obtenir des cristaux stables de ribosomes actifs.
Après avoir produit les cristaux, nous savions que nous allions faire face à une autre difficulté. Le faisceau de rayons X que nous avons dû utiliser pour déchiffrer la structure du ribosome était de très haute énergie (produit dans des accélérateurs géants, par exemple, à Grenoble, Stanford ou APS) au point d’affecter les liaisons chimiques entre les atomes des cristaux de ribosomes et provoquer leur déplacement. Cela signifie que lorsque nous irradions le cristal pour collecter des données sur sa structure, nous le détruisions. En pratique, nous n’aurions donc pas pu mesurer plus de quelques pourcents de l’information nécessaire pour déterminer la structure du ribosome. Nous savions que nous devions trouver un moyen de collecter suffisamment de données à partir du ribosome cristallisé avant qu’il ne soit détruit. Nous avons donc essayé de fabriquer des cristaux qui dureraient plus longtemps afin de pouvoir faire plus de mesures. Nous avons mis au point une méthode pour réduire les dommages causés par le rayonnement X en congelant rapidement les cristaux à très basse température (environ -200°C, en utilisant de l’azote liquide). Ces conditions qui empêchent la formation de cristaux de glace dans le liquide entourant le ribosome cristallisé visent à minimiser la capacité des atomes à se déplacer après que leurs liaisons aient été rompues par le rayonnement intense. L’utilisation de mélanges particuliers de solvants spéciaux nous a également permis de minimiser la propagation des dommages causés par le rayonnement. Nous avons alors pu mesurer la diffraction de petites zones de cristaux refroidis, en minimisant les dommages pendant les mesures et en épargnant les zones du cristal qui n’avaient pas encore été exposées au rayonnement. En utilisant cette méthode et d’autres procédures expérimentales développées des années plus tôt à l’Institut Weizmann des Sciences, nous avons réussi à maintenir les capacités de diffraction des cristaux du ribosome pendant une longue période, et finalement, après 20 ans, à en établir la structure et à comprendre son fonctionnement.
Tout au long de notre projet, de nombreux scientifiques pensaient que c’était impossible. Des scientifiques bien connus se sont même moqués de nous pour le défi colossal que nous essayions de relever. Mais grâce à notre détermination et à notre créativité, nous avons réussi à accomplir notre mission en recherchant constamment des solutions originales. En fait, malgré la méfiance du monde scientifique envers nos capacités et un grand mépris pour notre procédure de refroidissement, toute la bio-cristallographie l’a adoptée en quelques mois. Notre succès remarquable a également conduit à une application médicale importante, que tu découvriras dans le paragraphe suivant.
Lutter pour la santé – comment neutraliser les bactéries ?
Comme mentionné précédemment, le ribosome est un composant très important de la cellule. Lorsque la fonction du ribosome est significativement altérée, la cellule cesse de produire des protéines et ne peut pas continuer à vivre. Cela est également vrai pour les bactéries. Les ribosomes bactériens fonctionnent de manière similaire aux ribosomes humains, mais leurs structures sont légèrement, mais significativement différentes de celles des ribosomes humains. Ces différences nous permettent d’utiliser des antibiotiques qui nuisent aux bactéries sans affecter les patients qui prennent les antibiotiques.
Il existe plusieurs familles d’antibiotiques. Les antibiotiques naturels sont produits par certaines bactéries comme armes dans les ≪ guerres ≫ entre différentes bactéries qui luttent pour utiliser les mêmes ressources. Ils attaquent d’autres bactéries en paralysant un mécanisme essentiel à l’activité bactérienne. L’une de ces cibles est le ribosome. Bien que le ribosome soit énorme par rapport à la taille de l’antibiotique, de nombreux antibiotiques se lient et bloquent l’un des sites actifs des ribosomes : ils peuvent inactiver le PTC ou bloquer le NPET dans la grande sous-unité ribosomique, empêchant ainsi la croissance de la chaîne protéique naissante (Figure 4) en interrompant son allongement. Une autre stratégie est de bloquer le mouvement de l’ARNm dans la petite sous-unité ribosomique, ce qui empêche l’ARNm d’être correctement lu. D’autres antibiotiques ciblent le site A de la petite sous-unité ribosomique et empêchent l’ARNt de se lier au ribosome. Lorsque cela se produit, l’acide aminé ne peut pas se lier à l’acide aminé suivant dans la chaîne [4]. Clique ici pour en vidéo comment les antibiotiques peuvent perturber la fonction des ribosomes bactériens.
Les résultats de nos recherches ont donc permis des applications importantes dans le domaine de la médecine. Mais tu as peut-être entendu dire que certaines bactéries pathogènes ont développé une résistance à certains antibiotiques, ce qui les rend encore plus dangereuses. Comprendre la structure et la fonction du ribosome nous a permis de mieux comprendre comment fonctionnent différents antibiotiques et comment les bactéries ont développé des mécanismes de résistance, fournissant ainsi des idées sur la façon de lutter contre cette résistance. Ces perspectives justifient notre persévérance et le combat que nous avons mené pendant des dizaines d’années pour élucider la structure et la fonction du ribosome. Dans ce contexte, j’aimerais partager avec toi certaines leçons que j’ai retenues de cette recherche.
Recommandation pour les jeunes esprits
Ma principale recommandation est de garder ta curiosité. Si tu essaies d’apporter de nouvelles informations à un domaine scientifique qui te passionne énormément, tu pourrais rencontrer des problèmes pour trouver des soutiens, surtout au début. Certains te diront que tu n’as aucune chance de réussir, d’autres que tu fais fausse route dans ta recherche, et ils pourraient même prétendre que tu gaspilles l’argent public. Je te recommande de te concentrer sur ta curiosité – d’être fort et de croire en toi-même si tu n’as pas encore atteint ton objectif. Si tu approfondis le problème qui t’intéresse sur une base logique, tu augmenteras tes chances de trouver la solution. Mon but était de connaître la structure du ribosome. Tout au long du processus, j’ai cru que, même en cas d’échec, j’apporterai des progrès scientifiques significatifs en cours de route. Avant même que la structure du ribosome ne soit établie, mes recherches ont conduit à mettre au point de nouvelles méthodes en cristallographie. Elles ont permis d’élucider des dizaines de milliers de structures jusque-là inconnues et ont contribué à améliorer notre compréhension de nombreux processus biologiques.
Un autre aspect que j’aimerais partager avec toi est que plus le chemin est difficile, plus la satisfaction finale est grande. Sois donc déterminé et travaille pour surmonter les difficultés qui se dressent sur ton chemin, confiant qu’une grande satisfaction t’attendra au final.
Contributions à La Version Française
TRADUCTEUR: Jean-Marie Clément (Association Jeunes Francophones et la Science)
ÉDITEUR: Catherine Braun-Breton (Association Jeunes Francophones et la Science)
Glossaire
ADN: ↑ Support de l’information génétique transmis par les parents à leur progéniture. Il contient les instructions, appelés gènes, pour produire les composants cellulaires. L’ADN est constitué de l’enchaînement de nucléotides.
Protéines: ↑ Molécules ≪ ouvrières ≫, assurant la plupart des tâches dans la cellule et l’organisme en général, comme la production de mouvement (activité musculaire), le transport de l’oxygène et du dioxyde de carbone (respiration), la structure de l’organisme (peau, tissus conjonctifs) et sa protection (système immunitaire).
Acides Aminés: ↑ Éléments constitutifs des protéines (il y en a 20 différents) liés entre eux par une liaison peptidique. Une protéine de taille moyenne se compose d’une chaîne de 150 à 300 acides aminés.
Liaison Peptidique: ↑ Liaison chimique qui relie deux acides aminés dans une protéine.
ARN Messager (ARNm): ↑ Type d’ARN qui copie et transfert l’information génétique de l’ADN au ribosome pour la synthèse d’une protéine. Il permet au ribosome d’assembler une chaîne d’acides aminés dans le bon ordre en fonction du code génétique. Il est détruit plus ou moins rapidement une fois traduit.
Transcription: ↑ Synthèse d’une molécule d’ARN messager, copie d’une partie de l’ADN (gène) contenant les instructions pour la production d’une protéine. L’ADN est essentiel et ne peut pas être remplacé ; la cellule a différents mécanismes pour le préserver, y compris le mécanisme de transcription.
Ribosome: ↑ Organite cellulaire fabriquant les protéines. Il se compose de deux sous-unités (une petite et une grande) composées de nombreuses protéines et d’ARN ribosomique. La petite sous-unité assure le décodage du code génétique porté par l’ARN messager ; la grande sous-unité assure la synthèse des protéines selon les instructions du matériel génétique.
Traduction: ↑ Deuxième étape de la synthèse des protéines, dans laquelle l’information génétique portée par l’ARN messager est utilisée par le ribosome pour assembler une chaîne d’acides aminés. Au fur et à mesure que les acides aminés sont ajoutés à la chaîne en croissance, la protéine avance dans son tunnel vers la sortie du ribosome et se replie dans sa structure tridimensionnelle finale.
ARN Ribosomique (ARNr): ↑ Trois types d’ARN formant le cœur structurel et catalytique du ribosome. Environ les deux tiers de la masse du ribosome sont des ARNr, l’autre tiers correspond aux protéines ribosomales.
ARN de Transfert (ARNt): ↑ Type particulier d’ARN portant un acide aminé spécifique et se plaçant dans le ribosome pendant la traduction. Chaque ARNt porte une séquence anticodon qui se lie à un codon complémentaire sur l’ARNm, ce qui permet de placer l’acide aminé en bonne position pour allonger la chaîne protéique en formation.
Cristallographie: ↑ Technique utilisée pour déterminer la structure tridimensionnelle des molécules, y compris les protéines et les acides nucléiques. Il s’agit de fabriquer des cristaux de la molécule et de les irradier avec un faisceau de rayons X pour produire un motif de diffraction qui peut être utilisé pour calculer les positions des atomes dans les molécules dans la cellule unitaire. Cette technique est utilisée pour comprendre la fonction et les interactions des protéines.
Cristal: ↑ Matériau solide avec un motif répétitif d’atomes ou de molécules. Ce motif confère aux cristaux des propriétés spéciales, telles que la capacité de diffracter la lumière et de générer de l’électricité. Les scientifiques utilisent des cristaux pour étudier la forme et les liaisons internes de molécules telles que les métaux, les composés organiques, les protéines et l’ADN qui sont trop petits pour être vus au microscope optique.
Conflit d’intérêts
Les auteurs déclarent que les travaux de recherche ont été menés en l’absence de toute relation commerciale ou financière pouvant être interprétée comme un potentiel conflit d’intérêts.
Remerciements
Nous tenons à remercier Zehava Cohen d’avoir fourni les figures 1, 2 et 5.
Références
[1] ↑ Schluenzen, F., Tocilj, A., Zarivach, R., Harms, J., Gluehmann, M., Janell, D., et al. 2000. Structure of functionally activated small ribosomal subunit at 3.3 Å resolution. Cell 102:615–23. doi: 10.1016/S0092-8674(00)00084-2
[2] ↑ Zhou, X, Liao, W-J, Liao, J-M, Liao, P, Lu, H. 2015. Ribosomal proteins: functions beyond the ribosome. Mol. Cell. Biol. 7:92–104. doi: 10.1093/jmcb/mjv014
[3] ↑ Bashan, A, Agmon, I, Zarivach, R, Schluenzen, F, Harms, J, Berisio, R, et al. 2003. Structural basis of the ribosomal machinery for peptide bond formation, translocation, and nascent chain progression. Mol. Cell 11:91–102. doi: 10.1016/S1097-2765(03)00009-1
[4] ↑ Schlünzen, F., Zarivach, R., Harms, J., Bashan, A., Tocilj, A., Albrecht, R., et al. 2001 Structural basis for the interaction of antibiotics with the peptidyl transferase center in eubacteria. Nature 413:814–21. doi: 10.1038/35101544
